| ← Февраль 2012 → | ||||||
|
1
|
2
|
3
|
4
|
5
|
||
|---|---|---|---|---|---|---|
|
6
|
7
|
9
|
12
|
|||
|
13
|
14
|
15
|
16
|
17
|
18
|
19
|
|
20
|
21
|
22
|
24
|
25
|
||
|
28
|
29
|
|||||
За последние 60 дней 36 выпусков (4-5 раз в неделю)
Сайт рассылки:
http://nanoworld.narod.ru
Открыта:
07-10-2005
Статистика
-1 за неделю
Нанотех против Пикотех, что сабля против пулемёта...
|
Выпуск 284 Лаборатория Наномир Когда реальность открывает тайны, Нанотех против Пикотех, что сабля против пулемёта... Начинается с отметки 7 минут 20 секунд. Длительность ~2 минуты. Смотреть всем обязательно! Проверяем альтернативные пикотехнологические модели аминокислот. Victoria: Александр Юрьевич, а Вы могли бы написать скрипт для такого вида шаблона аминокислоты:
Кушелев: Первые четыре атома: N = Hedra family:1 scalep:100 scaleq:100 scaler:100 radius:0.92 pos:[0,0,0] p:0.4 wirecolor:[0,0,250]; Converttomesh N
В принципе из этой модели уже можно пытаться собирать альфа-спираль и другие типы структур (пи-спираль, бета-спираль, 310-спираль)... Victoria: А.Ю., в отличие от остальных атомов аминокислоты (N, CA, C) кислород (О) Вы не прописали в скрипте заново, а скопировали с имеющегося октаэдра для углерода С. Хотелось бы поправить его радиус, уменьшим до 0.88 и установить координаты его центра. Вы многократно использовали в скрипте команды move и rotate поэтому я не пойму, где конечные координаты центра красного октаэдра. Кушелев: Координаты можно измерить в параллельных проекциях 3DS Max. Victoria: Модель правой альфа спирали из последней модели шаблона для аминокислот: Кушелев: Уважаемая Виктория! К сожалению, в Вашей модели альфа-спирали на один виток приходится не 3.6 аминокислотных остатка, а меньше трёх, т.е. ~2/5 Попытайтесь ещё раз сделать модель альфа-спирали. Виктория: Не может быть. А Вы не ошиблись? Вертикальные водородные связи образуются в этой модели как раз между кислородом первого а.о. и азотом четвёртого. Это значит, что всё-таки 3,5 - 4 а.о. на виток. Кушелев: Водородные связи - дело десятое. Важно, на сколько аминокислотных остатков поворачивается спираль, совмещаясь со своим прежним положением. В Вашей модели явно меньше 2.5 аминокислотных остатков на виток, а экспериментальное значение 3.6 ... 3.7 Вы однозначно проделали большую работу, провели ряд модельных экспериментов. Фактически Вы получили доказательство того, что от модели альфа-спирали из современного учебника неизбежен переход к кольцегранной пикотехнологической модели. Строение аминокислот и аминокислотных остатков имеют особенности, характерные для полуацеталей, в которых углы между направлениями на центры атомов не просто отличаются от тетраэдрических, а могут плавно меняться.   Альфа-спираль из многогранников никак не получается, т.е. поворот на каждом аминокислотном остатке получается существенно больше, чем на угол 360/3.6=100 градусов. В частности, в этой модели поворот на каждом аминокислотном остатке происходит на угол, превышающий 150 градусов: Можно сделать, как в учебнике, т.е. уменьшить этот угол, но при этом грани многогранников уже не сойдутся. Более того, из учебника органической химии известно, что углы в альфа-спирали не тетраэдрические: Модель группы R-COOH из учебника. Пикотехнологическая модель группы -COO- Все видят, что атомы лежат в одной плоскости? 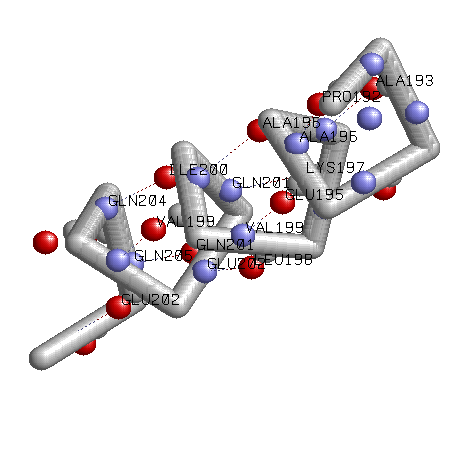 На этой модели альфа-спирали (из учебника) видно, что атомы группы СО, атом Сальфа и атом азота следующего аминокислотного остатка лежат в одной плоскости. Единственная неточность - радикалы перепутаны с атомами водорода. Отсюда и разговор об L-форме аминокислот. Достаточно переставить радикалы,
и форма L1 перейдёт в D2 4 типа спиралей (альфа-, 310-, бета- и пи-) можно посмотреть в 205-ом выпуске рассылки "Новости лаборатории Наномир": http://nanoworld88.narod.ru/data/205.htm В частности, пикотехнологическая
модель альфа-спирали:http://video.yandex.ru/users/nanoworld/view/2352/#hq
    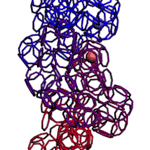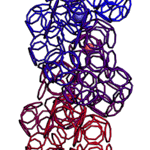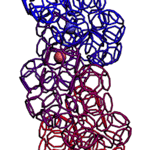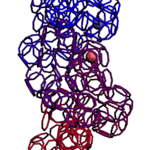  Пи-спираль и альфа-спираль (пикотехнологические модели). Подробнее: http://nanoworld88.narod.ru/data/221.htm Victoria: Примерно такая альфа-спираль:
Я вижу в Вашей модели разные композиционные углы. Пикантная идея, которая заключается в том, что по коду альфа-спирали, например, ggc ggc ggc ggc ggc ggc ggc ggc
ggc ggc ggc ggc, рибосома ставит аминокислоты в растущую белковую цепь под разными композиционными углами, меняя их таким образом, чтобы в среднем получалось 3.6 ... 3.7 аминокислотных остатков на виток альфа-спирали. Я правильно понял Вашу идею? Если так, то давайте её проверим. Victoria: Вообще то "композиционные углы" планировались одинаковыми . Но если в ручном режиме я присоединила аминокислоты не совсем точно, то давайте напишем скрипт, по которому аминокислоты будут единообразно присоединяться друг к другу, водородные связи будут одинаковыми и мы сможем посчитать количество а.о. на виток в такой модели альфа-спирали. Кушелев: Я уже пробовал. Одинаковые тетраэдрические углы не дают параметров альфа-спирали ни при каких комбинациях. Поэтому и удивился, когда Вы показали последнюю модель. Кушелев: Мы с Дмитрием Кожевниковым ещё в 1992-ом году поняли, что альфа-спираль из тетраэдрических углов не составить. Я ему сразу сказал, что делать модель аминокислоты - практически безнадёжное дело. Но он парень дотошный и настойчивый. Он достал толстенный учебник по органической химии и нашёл там структурную схему полуацетали, где связи между атомами имеют ту же природу, что и в аминокислоте. Я ему говорил: "Брось ты это безнадёжное дело!", а он мне и отвечает: "Раз модель бензола и модели нуклеотидов получились, хотя ты тоже говорил "безнадёжное дело", то может быть и модель аминокислоты получится". Ну и короче, добился того, что я тоже начал вместе с ним делать модель аминокислоты. С очередной попытки мы получили эту модель полуацетали: или ацетали. Я уже точно не помню название... Потом была сделана модель молекулы мочевины Далее к этой модели была добавлена группа азота: Так и появилась первая пикотехнологическая модель аминокислоты... Мы выбрали L-форму, согласно учебнику. Позднее я понял, что радикал нужно переставить. При этом L1-форма превратилась в D2-форму, которую я заменил на зеркально-симметричную, т.е. L2-форму. Из неё получается зеркальная спираль. Теперь это недоразумение устранено, и правая альфа-спираль строится из D2-форм аминокислотных остатков.
Виктория: Мой вариант моделей аминокислот:
Кушелев: Я правильно понял, что осталось одно несущественное отличие, радикал. Переставим? Кстати, на Вашей иллюстрации упрощённая модель не соответствует кольцегранной. Причём по угловым величинам сильно не соответствует. И по линейным величинам тоже... А самое большое несоответствие Вашей и моей модели конечно же в положении радикала. Я бы сказал "радикальное несоответствие". Но в случае глицина этого радикального несоответствия нет, поэтому давайте разберёмся, есть ли различия между нашими моделями глицина ? Модель B и C с Вашей иллюстрации (если не считать радикала) в точности совпадают с моей, т.к. они из неё и сделаны путём перестановки радикала А раз совпадают кольцегранные, значит должны совпасть и упрощённые, т.е. многогранные. А раз совпадают упрощённые, значит совпадает и скрипт. Единственное отличие, которое осталось - это другое расположение радикала. Тут можно проверить на уровне белков, где существенно положение радикалов. Попробуем проверить на пептидах с дисульфидными мостиками... фрагмент препроокситоцина, состоящий из 9 аминокислотных остатков. После сборки рибосомой этот фрагмент модифицируется и вырезается из белка-предшественника. Есть похожий белок, вазопрессин. Композиционный код фрагмента пропревазопрессина: 1,1,1,1,1,1,4,1,1
dne-файл: 60FT Вазопрессин / vasopressin получается из этого фрагмента путём модификации, т.е. изменения композиционных углов после сборки белка-предшественника рибосомой.
Фрагмент А-цепи инсулина, который должен быть замкнут через дисульфидный мостик. Однако, для этого нужно изменить один или несколько композиционных углов после сборки А-цепи рибосомой. Интересно разобраться, какие углы нужно изменить, т.е. какие аминокислотные остатки повернуть, чтобы цикл замкнулся через дисульфидный мостик? Я припоминаю, что приблизительно такая же последовательность композиционных углов у меня получалась в пластмассовой модели цикла окситоцина. Именно модель окситоцина помогла мне понять, что кроме альфа-спирали, 310-спирали и бета-спирали есть ещё одна спираль, которую я назвал кси-спиралью, но оказывается у неё уже есть общепринятое название, пи-спираль. Сейчас я подберу композиционный код, чтобы замкнуть цикл из 6 аминокислотных остатков. При этом в случае изменения позиции радикалов цикл должен разомкнуться. Это и будет тот эксперимент, который позволяет выбрать один из двух положений радикалов...
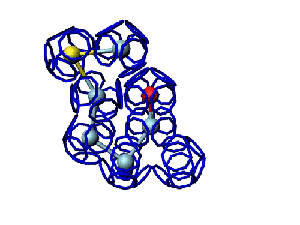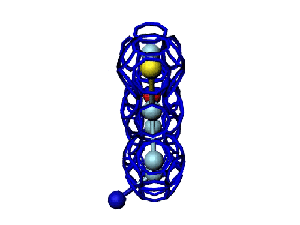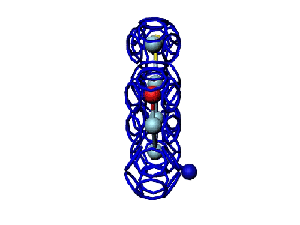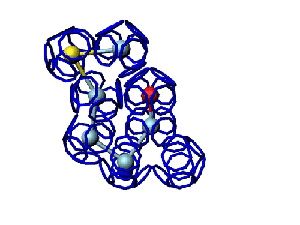
Начальная последовательность инсулина в этой зоне была: 333123 После модификации, когда композиционный код стал соответствовать: 333131, дисульфидный мостик (2 атома вверху, показанные жёлтым цветом) замкнулся. Теперь интересно проверить, возможно ли такое, если переставить радикалы по варианту Виктории Соколик? Уважаемая Виктория! Я выкладываю файл для 3DS Max, в котором радикалы цистеинов отделены от остальной части модели. http://nanoworld88.narod.ru/3DS/333131_d2.max Вы можете переставить их по своему усмотрению. Если получится альтернативная модель, в которой радикалы цистеинов дотянутся друг до друга, тогда Ваш вариант модели не будет противоречить результату модельного эксперимента. Victoria пишет:
Кушелев: А как быть с кодом gga ? Код этого фрагмента белка: tgt tgt ctt agc gga tgc
Так что Ваш вариант без модификации этого участка белка после рибосомальной сборки тоже не получается... Victoria пишет:
Кушелев: Так я об этом сразу написал. И в Вашей точно не получится, т.к. в нуклеотидной последовательности есть код бета-спирали gga. Но мы с Вами знаем, что после сборки на рибосоме белок-предшественник (преинсулин) модифицируется. Углы меняются. Пикотехнологическая модель показывает, что сразу цикл окситоцина, вазопрессина, инсулина... сделать не получается. Не влезают в цикл АСС-концы тРНК. Поэтому и приходится делать другую структуру (участок альфа-спирали, как в случае окситоцина и вазопрессина или участок пи-спирали, как в случае инсулина), из которой потом путём поворота двух (в случае инсулина) аминокислотных остатков получается цикл из 6 остатков, замкнутых через дисульфидный мостик. Модель окситоцинового цикла я сделал из пластмассовых колец ещё в 1992-ом году: Цикл окситоцина (без радикалов)
структура окситоцина из видеосюжета "Формы и механизмы молекул":http://video.mail.ru/bk/yaroslav2008/22/39.html Чтобы увидеть правильную форму пластмассовой модели, нужно смотреть на неё через зеркало. Неправильная модель - справа. Правильная - слева. Кушелев: Уважаемая Виктория! Я Вам открою ещё один секрет, который позволяет сделать однозначный выбор между положениями радикалов аминокислот. Это - модель метионина. Модели других аминокислот можно посмотреть в рассылке:http://nanoworld88.narod.ru/data/279.htmНапомню, что метионин гнёт альфа-спираль. Как он это делает? Он вставляет между собственным атомом кислорода группы СО и атомом азота аминокислотного остатка следующего витка дополнительный атом углерода, точнее группу CH3. Если радикал переставить, то он не дотянется... Идёт создание новой версии программы PikoTech,
которая уже начинает строить упрощённые 3D-структуры белков по композиционному коду... Структуру белка ещё
"плющит", но это - временные проблемы Ну вот, уже не плющит... *** Создан скрипт для MatLab, который строит пикотехнологическую модель стебля белковой молекулы по композиционному коду. % p = #() % TAtom p:[ 27.85, -19, -26.5] ch1:"N" ch2:"
" ch3:" ", function ColModel() for i = 1:1:473 plot3(Peptide1(1:473,1),Peptide1(1:473,2),Peptide1(1:473,3),'DisplayName','Peptide');hold on; fpdb=fopen('collagen.pdb','w'); str='ATOM
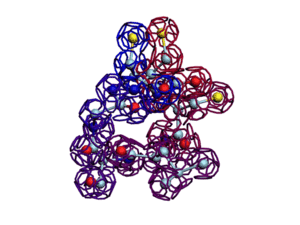
C GLY A 1 1.00 0.50 A N\r\n'; str='ATOM O GLY A 1 1.00 0.50 A N\r\n'; end В данном варианте скрипта входными данными является композиционный код белка коллагена, который выводит программа от Prosolver. В результате получаем pdb-файл стебля белка, т.е. без радикалов: Фрагмент pdb-файла, который выводит этот скрипт: ATOM 2 CA GLY A 1 -54.250-156.124 84.413 1.00 0.50 A N Технологии Гермеса... Гермес: добрый день!Укажите, пожайлуста, сферу ппрменения pdb-файла для чего он нужен и т. п. Это необходимо для продажи. Кушелев: pdb-файл содержит координаты атомов в молекуле белка. Нужен тем, кто использует структуру белка в своей работе. Фармацевты, генные инженеры, биотехнологи и все, кто связан с нанотехнологией. Пикотехнология - это развитие нанотехнологии, только в ~1000 раз точнее. Получают pdb-файлы с помощью рентгеноструктурного анализа или с помощью нейтронной дифракции. Для этого несколько месяцев выращивают кристалл из миллиардов миллиардов одинаковых молекул исследуемого белка. После этого просвечивают белковый кристалл рентгеном или нейтронами. Получают дифракционные картины в разных ракурсах и анализируют на суперкомпьютерах. Программа "Пикотех" позволяет смоделировать работу рибосомы и узнать структуру белка в ~1000 раз точнее и в миллион раз быстрее. Кроме того рентген и нейтроны пригодны лишь для 3% белков, которые кристаллизуются. Пикотех позволяет определать структуру белка независимо от способности кристаллизоваться. Ежегодно продаётся более 1000 pdb-файлов по средней цене ~10 000 евро. Все покупатели - наши потенциальные клиенты. Пригоден ли конкурс CASP для пикотехнологов? Victoria: На конкурсе вывесили новую задачу: >R0011 CD27L , , 81 residues Это кусок рецептора к цитокину CD27. Вот только организм они опять не указали, поэтому нуклеотидную последовательность найти будет почти невозможно. Кушелев: Нашёл другой цитокин: http://www.genome.jp/dbget-bin/www_bget?rno:500318 http://www.genscript.com/product_001/ge … 335.1.html 60FT
Композиционный код: 1,2,1,2,1,1,1,1,1,1,1,4,4,1,1,2,4,1,3,3,1,2,1,2,1,1,3,2,1,3,1,1,1,3,4,2,2,1,1,1,1,1,3,4,2,4,2,1,3,4, Cytocine (PyMOL, mesh) Если нужен pdb-файл, моглу выложить... Пикотехнологическая модель цитокина дикого кролика Готовим встречу с Даниилом Шостиным... Skype: 2012-02-20 [14:39:07] Alexey Komanyov: приветствую) Представим себе уменьшенную копию Стоунхэнджа, в которой все линейные размеры уменьшены в 10 раз. При этом объём каждого камня уменьшится в 10^3=1000 раз. Самый крупный камень в модели будет весить не 55 тонн, а 55 кг. Это - приблизительно средний вес студентки Таким образом, мы получили игрушечный Стоунхэндж, который меньше настоящего в 10 раз по "росту" и в 1000 раз по весу. А теперь попробуем сделать наборот, т.е. не уменьшим, а увеличим массу камней Стоунхэнджа в 1000 раз. Масса самого крупного камня (55 тонн) станет в 1000 раз больше, т.е. 55 000 тонн. Теперь вспомним, что масса одного из камней Храма Солнца в Ласпи (под Севастополем) превышает 70 000 тонн. Получается, что по сравнению с Храмом Солнца Стоунхэндж - игрушка "Летающие тарелки" бороздят ... мониторы  [12:43:24] В: Привет Саш! Посмотри видео про "летающую тарелку"http://www.youtube.com/watch?v=XwnhUbj0bf8
Пилот управляет внутри неё или это дистанционно управляемая для собирания драгметала тарелка? [12:48:01] Кушелев Александр Юрьевич: Похоже на компьютерную графику, но вполне может быть сделана по рассказам очевидцев... Скорее всего речь идёт о "тарелке"-роботе [12:48:35] В: И я так считаю Китайская клавиатура из ... Заповедника под голубым Солнцем
Цитата: "Несколько "человек" сидели за круглыми экранами..." Это - не Пандора! Это - Испания...
Фантастический лес. Кантабрии, Испания.
Лавандовые поля, Прованс, Франции Реальная относительность...
Кристаллические мыльные пузыри...  Этому пузырю понадобилось 15 минут, чтобы лопнуть. А знаете ли вы, что можно заморозить мыльный пузырь, просто бросив на него снежинку? Пузыри замерзают при -7 градусах по Цельсию. Снежинка соприкоснется
с пузырем и от нее во все стороны побегут бусинки льда. Причем тип кристаллизации будет такой же, как и у снежинки, то есть можно выбрать его заранее, отлавливая снежинки и рассматривая их на варежке... Из-за своей маленькой толщины пленка пузыря при этом остаётся пластичной, так что если уронить бывший мыльный пузырь на землю, то он не разобьется на множество ледяных осколков. Вместо этого на пузыре образуются вмятины, а если уронить шарик посильнее, он распадется на кусочки, которые тут же скрутятся в трубочки
НЕ ИЕЕМТ ЗАНЧНЕИЯ...
Приглашение к сотрудничеству для людей умеющих самостоятельно мыслить; не просто умных, а мудрых, которые чувствуют, где истинаЛаборатория Наномир готова к любому взаимовыгодному сотрудничеству. У нас есть сторонники как явные, которые помогают морально и материально, есть очень много пассивных наблюдателей, есть и ярые противники, которые используют любые методы и средства (аморальные и просто преступные), чтобы уничтожить работу лаборатории и дискредитировать ее. В одиночку
внедрить технологии, выводящие цивилизацию на новый уровень, невозможно. Благодаря поддержке множества заинтересованных людей проделана огромная работа. Ознакомиться с её результатами можно изучив материал рассылки "Новости лаборатории Наномир". Люди науки могут изучить научные труды. Вклад каждого не останется незамеченным в случае успеха в реализации научных проектов. Результаты совместной деятельности принадлежат участникам проекта пропорционально коэффициентам творческого и финансового участия. Основные разработки: Микроволновый источник энергии В 2011 году были куплены рубиновые шарики для эксперимента на сумму ~1000 долл. В результате было сделано научное открытие, проверена защита диэлектрических резонаторов от перенапряжения. В этом году, вероятно, можно будет создать микроволновую энергетику, т.к. удалось найти сырьё (рубин #8), из которого сделаны рубиновые шарики для эксперимента в Дубне. Параллельно началась серия экспериментов на ускорителе "B" в диапазоне 20 ... 140 ГГц. "Эликсир вечной молодости" 28 сентября 2011г. в институте геронтологии (г.Киев) начался эксперимент по созданию "эликсира вечной молодости". Благодаря первому взносу (в размере 500 долларов) Золдракса и поддержке других соинвесторов. Продолжаются переговоры с потенциальными инвесторами по поводу финансирования этого проекта. Программа исследований Презентация Пикотехнология Созданы первые версии пикотехнологии (выпуски рассылки 212 270 271 272 ), с помощью которой Александр Кушелев и Виктория Соколик сделали более10 научных открытий. Виктория Соколик: Уважаемые коллеги, Вашему вниманию предоставляется услуга -- моделирование 2D и 3D структуры любого белка без ограничений в его размере и степени изученности с помощью программного обеспечения, базирующемся на принципиально новом подходе декодирования нуклеотидной последовательности, детерминирующей данный белок. Всё, что необходимо от заказчика, это нуклеотидная последовательность мРНК интересующего его белка (или код этой нуклеотидной последовательности в EMBL, или хотя бы код самого белка в PDB). В течение 1-3 суток мы готовы предоставить Вам схему вторичной структуры заказанного белка (2D), модель его пространственной структуры (3D) в виртуальном пространстве, а также файл .pdb с координатами каждого атома белка. Файл .pdb может быть использован по аналогии с файлами закристаллизованных белков из PDB банка для дальнейшего конформационного анализа белка методами молекулярной динамики с учётом физико-химической специфики микроокружения белка или его взаимодействия с лигандами. Таким образом, Вы сможете максимально быстро удобным для Вас способом (по электронной почте, на сайте либо на электронном носителе) получить информацию о структурном шаблоне Вашего белка. Первые 10 заказов -- бесплатно . Цена следующих заказов -- договорная. Сотрудничество может быть различным: - участие в научных дискуссиях на форуме (конструктивное) - совместное создание коммерческого продукта - поиск инвесторов - выступить менеджером по продаже готовых коммерческих продуктов - конструктивные предложения по продвижению идей лаборатории Наномир - содействие в проведении экспериментов
и т.п. - написание совместных научных статей и т.п. - материальный вклад (денежный или обеспечение оборудованием и материалами)
Пожалуйста, сообщайте о своем вкладе, чтобы мы зачли Вас как партнера лаборатории Наномир. +7-926-5101703, +7-903-2003424, +7-916-8265031, Skype: Kushelev2009, mail: kushelev2011@yandex.ru веб-мани: WM-кошелек R426964799301
|
| В избранное | ||











































.jpg)
.jpg)



