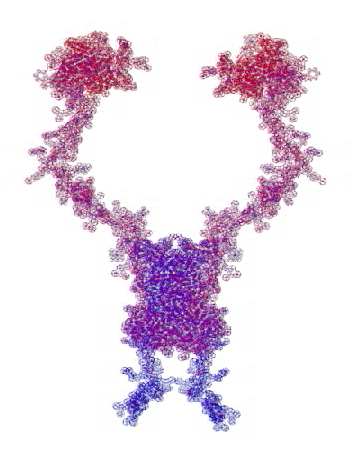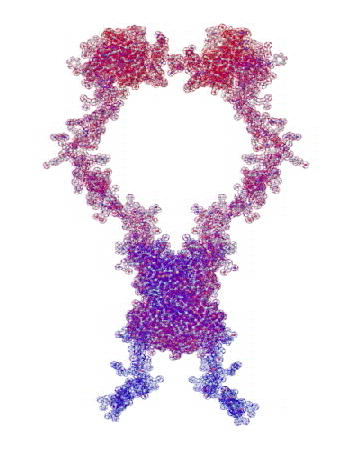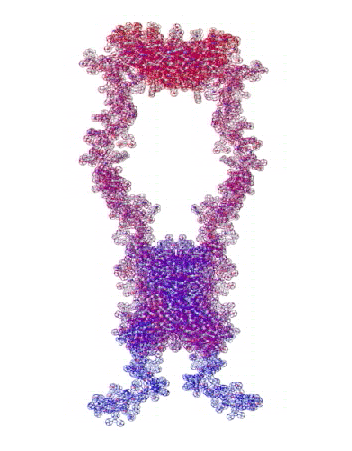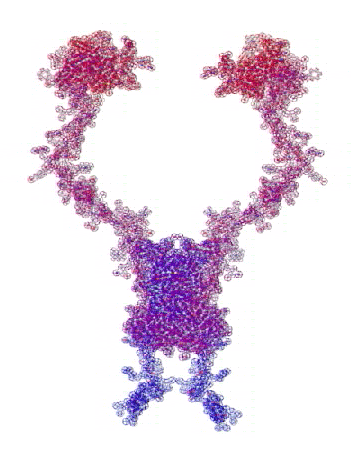| ← Апрель 2022 → | ||||||
|
2
|
3
|
|||||
|---|---|---|---|---|---|---|
|
4
|
5
|
6
|
7
|
8
|
9
|
10
|
|
11
|
12
|
14
|
15
|
16
|
||
|
18
|
19
|
20
|
21
|
22
|
23
|
24
|
|
25
|
26
|
27
|
28
|
29
|
||
За последние 60 дней 36 выпусков (4-5 раз в неделю)
Сайт рассылки:
http://nanoworld.narod.ru
Открыта:
07-10-2005
Статистика
-1 за неделю
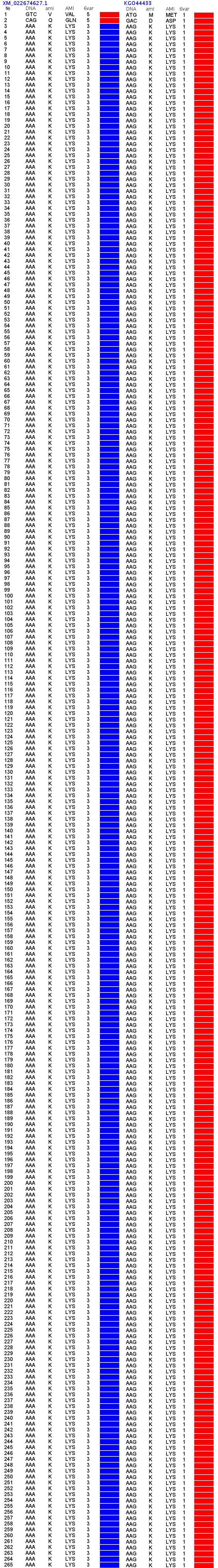
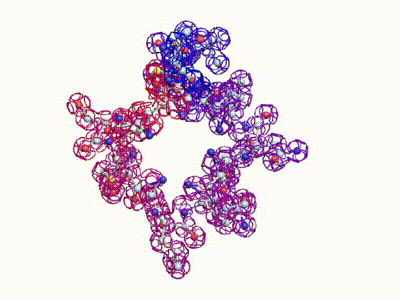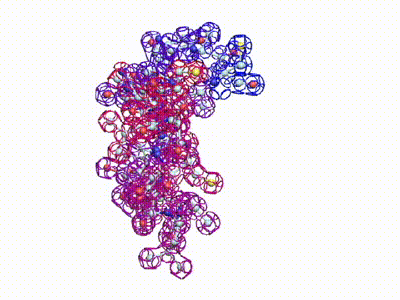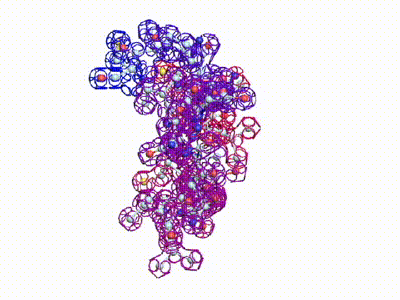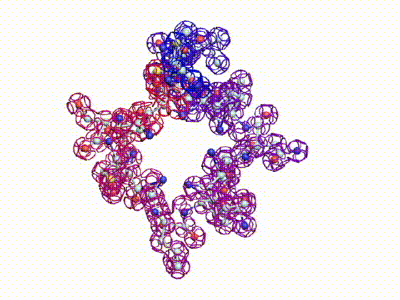
807 Почему Институт белка не может воспользоваться пикотехнологией 2D?
|
| В избранное | ||