| ← Декабрь 2017 → | ||||||
|
2
|
3
|
|||||
|---|---|---|---|---|---|---|
|
5
|
6
|
7
|
8
|
9
|
10
|
|
|
12
|
13
|
14
|
15
|
|||
|
18
|
19
|
21
|
22
|
23
|
24
|
|
|
25
|
26
|
27
|
28
|
29
|
||
За последние 60 дней 56 выпусков (примерно раз день)
Сайт рассылки:
http://nanoworld.narod.ru
Открыта:
07-10-2005
Статистика
+1 за неделю
638 В гостях у Кушелева Глобальная Волна
|
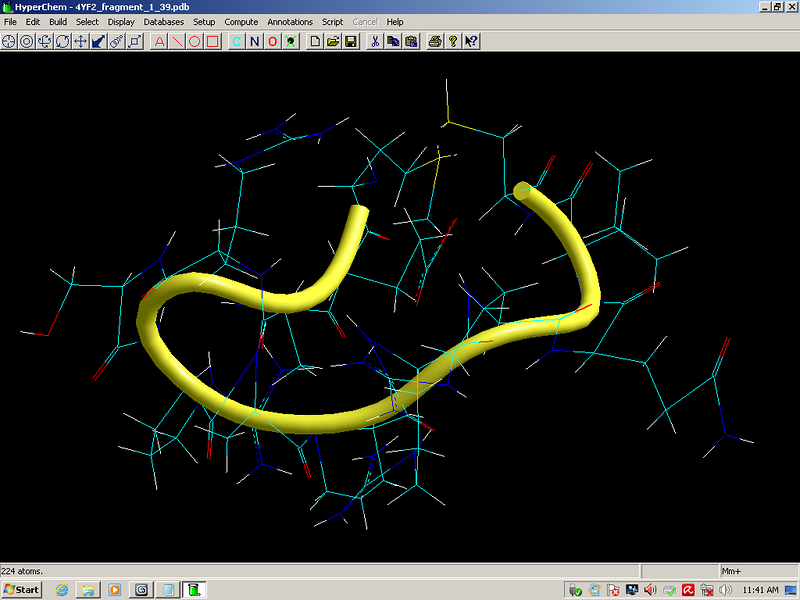
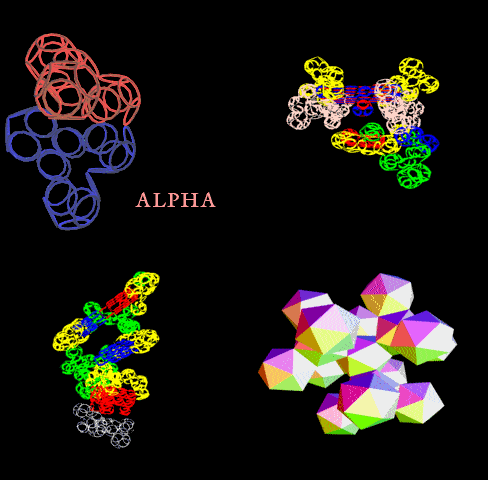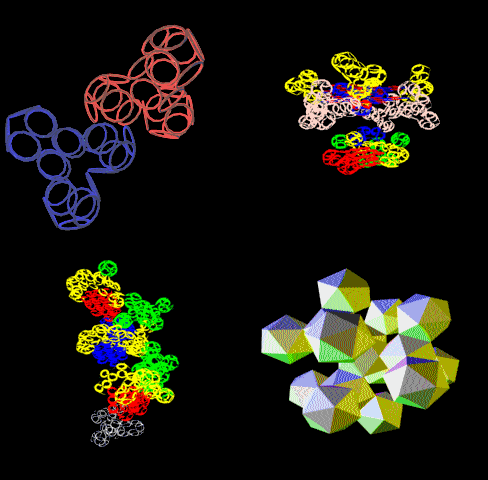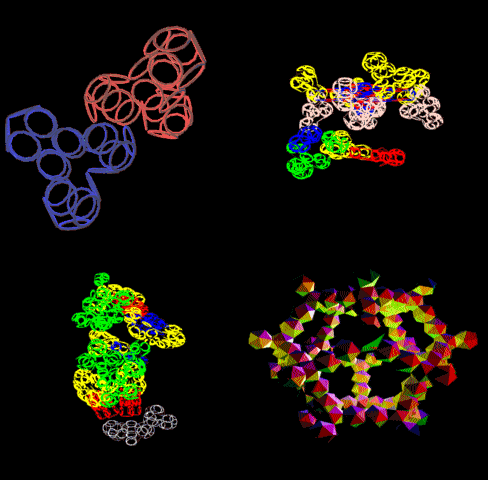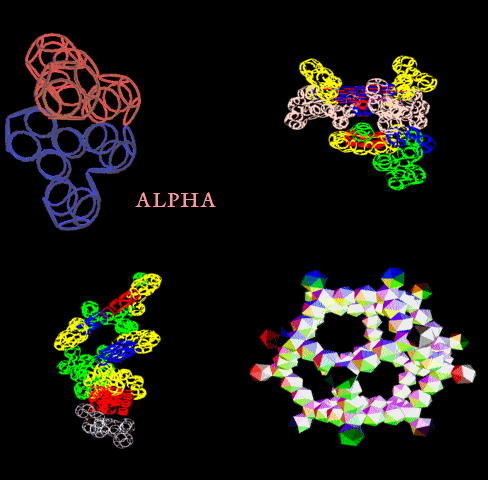
Выпуск 638 Геномная версия программы Пикотех позволяет определять структуры белков в миллиард раз быстрее рентгеноструктурного анализа. Лаборатория Наномир Когда реальность открывает тайны, В гостях у Кушелева Глобальная Волна https://www.youtube.com/watch?v=uAZpTdTGH4g
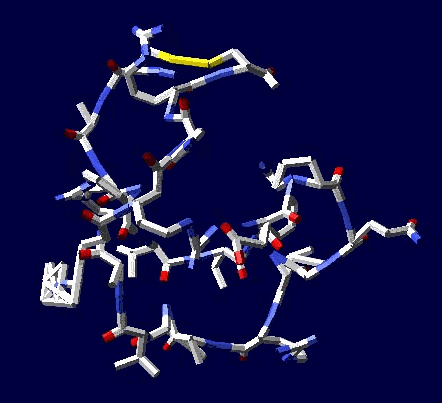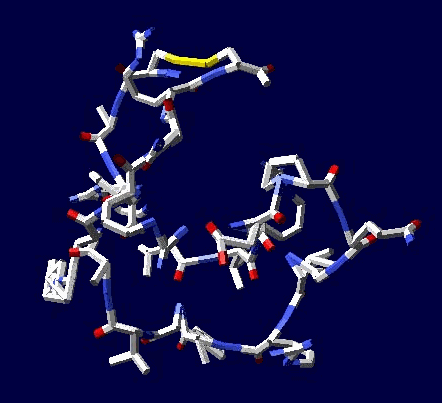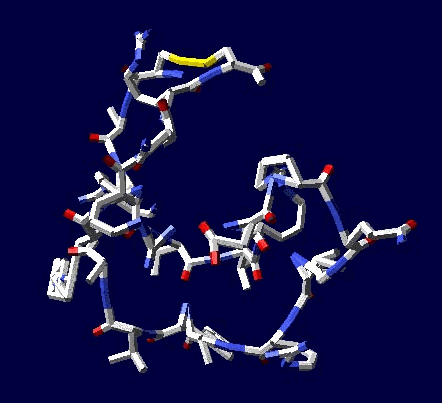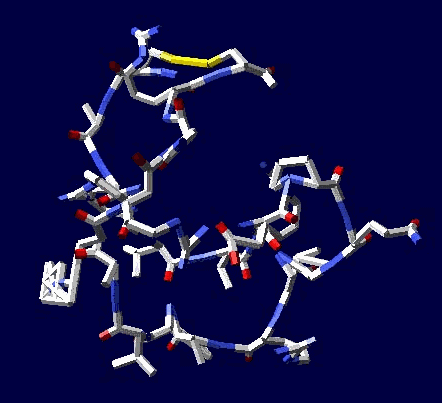
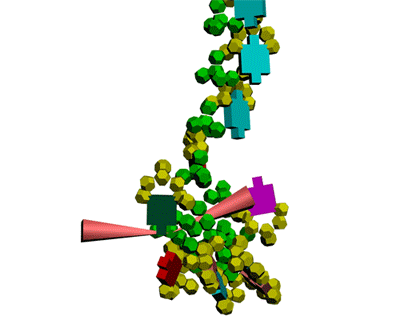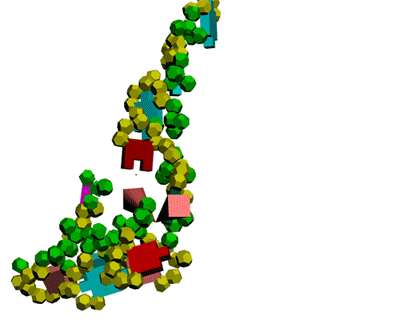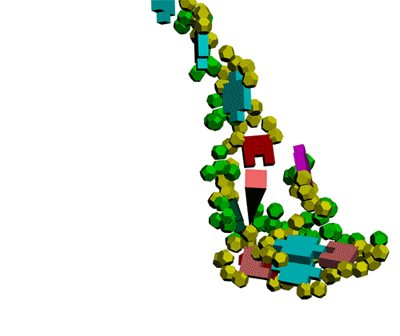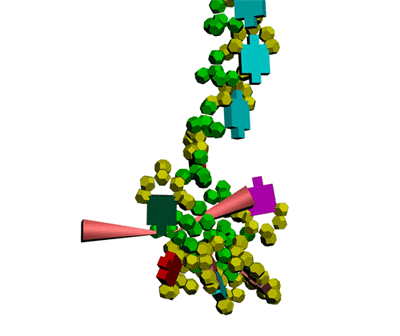
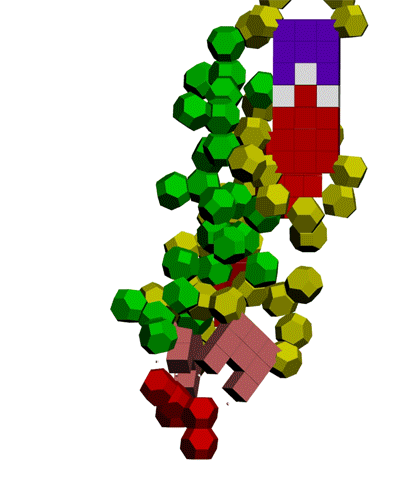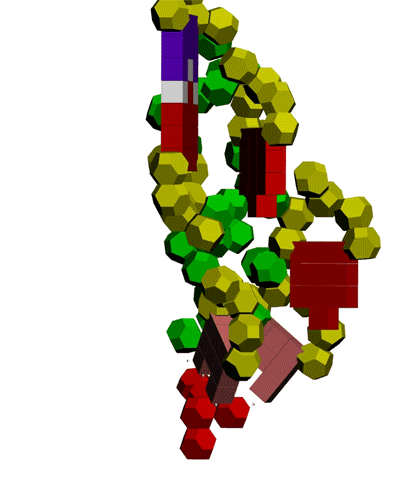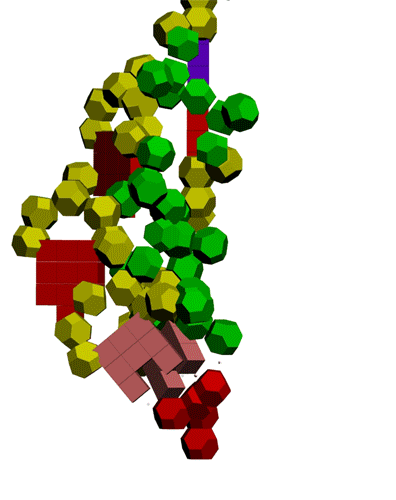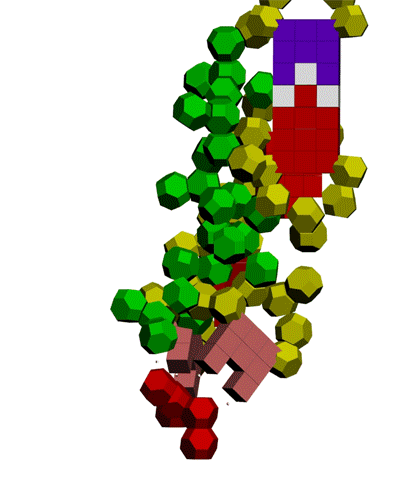
Многослойные белковые спирали и мезо-механизмы Многослойные белковые спирали и мезо-механизмы Рекорды сверхдлинных спиралей белковых молекул Online service PROTEIN PICOTECHNOLOGY Skype, 2017-12-17: [2:20:01] Кушелев Александр Юрьевич: Сегодня удалось сделать крутое научное открытие: http://nanoworld.org.ru/topic/1881/ [2:20:09] Кушелев Александр Юрьевич: Поздравляю! Это наша удача. [9:29:57] Кушелев Александр Юрьевич: "Сам себя не похвалишь, никто не похвалит". А ты же зарегистрирован на форуме. Давай обсудим нашу удачу. В чём проблема?
Развёрнуто:
Кстати, мне уже попадались аналогичные и даже "более такие же", "более чем" структуры:
Развернуто:
Эта структура вообще может быть Т-конструкцией из двухслойной спирали. Сначала строится верхняя черта Т из 310-спирали длиной 38 аминокислотных остатков. Затем поверх нее укладывается второй слой (пи-спираль длиной 17 АО), покрывая процентов 20...25 310-спирали. Потом идёт шпилька из 310-спирали длиной 8 АО (примерно 0.21 от "верхней черты"). Далее обратным ходом на шпильку укладывается 19 АО пи-спирали. После этого строится 28 АО альфа-спирали, которая может идти под любым углом, т.е. от нуля до 180 к первоначальной "черте" или к шпильке. Кстати, шпилька тоже может быть расположена под любым углом к "черте". Далее идёт 310-спираль длиной 45 АО и альфа-спираль длиной 53 АО. Последние три участка - это сверхдлинная прямая спираль (28+45+53 АО), которая тоньше в средней части. Наконец, идёт пи-спираль, например, поверх альфа-спрали и какой-то хитрый узел. Всё это может иметь форму рогатки из двухслойной пи-альфа-спирали на всех трех концах. Может быть и сравнительно низкочастотной резонансной системой, похожий на U-образный музыкальный камертон. С этим интересно было бы разобраться... ***
Развернуто:
Этот белок тоже может состоять из двухслойных спиралей, но верхний слой может быть образован не фундаментальной, а программной спиралью. У меня и раньше закрадывалось предположение, что программная 35-спираль может идти верхним слоем по отношению даже к пи-спирали, не говоря о более тонких спиралях.
А этот белок содержит длинные участки 310-альфа-спиралей, пи-спиралей и программных 35-спиралей. Я не удивлюсь, если в его структуре попадётся не только участок двухслойной, но и участок трехслойной альфа-пи-35-спирали.
Здесь мы тоже видим участок 310-спирали, за ним сразу пи-спираль, а за ним 35-спираль. Это может быть трехслойный участок 310-пи-35-спирали. А может и нет. Надо проверять...
Если это - система противонаправленных альфа- и бета-спиральных участков, образующих гигантскую трубу, то это:
Развернуто:
Может быть либо антинаправленными участками альфа-спиралей и двухслойных 310-пи-спиралей, образующих гигантскую трубу, либо рёберным многогранником, в частности, 6-угольной пирамидой, основание которой образовано альфа-спиральными участками, а наклонные ребра - двухслойными 310-пи-спиралями. При этом пирамида может быть лишь одним из крайних состояний некого манипулятора, который может развернуться в 6-конечную "звезду". Это нужно моделировать в 3D. Тогда может появиться определенность и ясность.
Это уже скорее всего гигантская труба из противонаправленных альфа-спиральных и двухслойных 310-пи-спиральных участков. Но тоже надо проверять.

Эти экзотические структуры могут оказаться очень интересными мезо-механизмами (что-то среднее между большой третичной и малой четвертичной структурой), включающими в себя многослойные спирали. Фолдинг есть, но на уровне третичной структуры белка. На уровне вторичной структуры его нет. Материал с форума лаборатории Наномир: aest: Если я не ошибаюсь, альфа спираль у Виктории получается в результате фолдинга.Кушелев: Совершенно верно! Рибосома по композиционному коду строит сначала 310-спираль (по Виктории):
  310-спираль . альфа-спираль
после этого мистический фолдинг (будем называть вещи своими именами) разрушает все водородные связи n ... (n+3), меняет все углы между аминокислотными остатками с 120 градусов (3 АО на виток) до 100 градусов (3.6 АО на виток), после чего создает все водородные связи заново. "Травматическая косметика" в двух словах Виктория Соколик: Фолдинг экспериментально показан, описаны белки-шапероны, принимающие в нем участие, Вам стоит только усвоить эту информацию и перестать быть таким категоричным: или кодирование, иди фолдинг. Учитесь у природы: в клетках И кодирование 3D-структуры, И её последующий фолдинг. Кушелев: Тут нужно уточнить, на каком уровне структуры существует фолдинг? Я утверждаю, что вторичная структура однозначно закодирована. Если код n(GGG), то из канала рибосомы выходит 310-спираль, которую никакой мистический фолдинг не переделает ни в альфа-спираль, ни в пи-спираль. Конечно, если сильно растянуть альфа-спираль, то она сначала перейдёт в бета-спираль с разрывом водородных связей, а после дальнейшего растяжения разорвутся и ковалентные связи. Но при снятии напряжения бета-спираль обратно в альфа-спираль не превращается. Другое дело - участки альфа-спирали, между которыми находится Pro. Они действительно могут работать как сустав:
Виктория Соколик: Углы композиции между аминокислотными остатками задаются внутри рибосомы, а вот водородные связи, дополнительно фиксирующие их взаимное расположение уже на выходе из неё. Кушелев: С чего бы это? Если аминокислота соединена с предыдущей, водородная связь образуется за миллисекунду:
Подробнее о протонной релаксации водородных связей: http://crm-en.ics.org.ru/uploads/kim3/crm09307.pdf
http://www.examen.ru/add/manual/school- … nformaczii Цитата: Скорость сборки одной молекулы белка, состоящего из 200-300 аминокислот, составляет 1-2 мин. Кушелев: Это значит, что очередная аминокислота устанавливается рибосомой примерно за 0.2 ... 0.6 сек. Пока будет присоединена следующая аминокислота, водородная связь 200 раз успеет образоваться. Виктория Соколик: В рибосоме водородные связи не образуются, только на выходе из неё за те же миллисекунды. Кушелев: С чего Вы это взяли? В канале рибосомы умещаются даже все радикалы альфа-спирали. Это значит, что там та же среда, что и за пределами рибосомы. Те же молекулы воды, протоны, образующие водородные связи. Как только присоединена очередная аминокислота (0.2 ... 0.6 сек), так уже через миллисекунду образуется водородная связь, т.е. до установки следующей аминокислоты. Другого варианта нет. Виктория Соколик: Вы уже заложили в свою таблицу композиционного кода и углы для альфа-спирали. Вам проще отстаивать эту ОШИБКУ Кушелев: Зачем отстаивать? Эксперимент показывает, что для замыкания дисульфидных мостиков "скрипач (фолдинг) не нужен"
Цикл, собранный по геометрическому алгоритму замыкается и без него:
Виктория Соколик: статистический анализ приведенный в монографии "Пикотехнология белков", на основе которого сделана моя таблица композиционного кода, не показал специфику кодирования альфа- и 3/10 спиралей РАЗНЫМИ кодонами. Вашей статистики на этот счет НЕТ, поэтому такое утверждение ГОЛОСЛОВНО и ненаучно его отстаивать. Кушелев: Если анализ не показал, то это не означает, что повторный анализ не покажет Мы с Вами знаем, что специалисты по рентгеноструктурному анализу редко отличают 310-спираль от альфа-спирали. Более того, за многие десятилетия существования РСА протяженные участки пи-спиралей вообще оказались незамеченными... А они есть!
Так что в следующей книге "Пикотехнология белков-2" у Вас есть возможность продемонстрировать и 6 фрагментов лизоцимов, замкнутых через дисульфидные мостики без фолдинга, и сверхдлинные фундаментальные и программные спирали белковых молекул, структура которых определена по таблице композиционного кода, где Ваши данные для альфа- и пи-спиралей полностью совпадают с моими. Вы ещё хотели показать экспериментальные данные, с которыми совпадают Ваши альфа- и пи-спирали длиной более 63 витков:
А если этих экспериментальных данных еще нет, то что мешает их получить? В институте белка мне сообщили, что определить структуру с помощью РСА могут в Курчатовском институте. Давайте вместе предложим им определить две структуры полилизина, закодированные триплетами n(AAG) и n(AAA). aest: лизоцим не является жесткой фигурой, подобной треугольнику, там вполне возможно согласованно поизменять углы без разрыва дисульфидного мостика, подобно тому, как это можно сделать в параллелограммеКушелев: Только он замкнулся через дисульфидный мостик без изменения углов. А это значит, что фолдинга нет на уровне вторичной структуры. Кстати, Вы можете "поизменять углы" и выложить на форум результат своего колдовства. Ваши "просто слова" против модельного эксперимента ровным счётом ничего не значат
Сначала измените согласованно углы, но не в параллелограмме, а в модели фрагмента лизоцима, а мы посмотрим, замкнётся ли он после этого... Виктория Соколик: Вы строите свою таблицу кодирования разновидностей спиралей опираясь на свои фантазии и отмахиваясь от результатов статистики.Кушелев: Вообще-то я сразу показал, как я строил свою таблицу композиционного генетического кода: http://www.nanoworld.org.ru/data/01/data/texts.rus/9920204.htm Я воспользовался данными РСА для наиболее изученных белков. При этом на пластмассовых моделях я получил параметры альфа-спирали, 310-спирали, пи-спирали и бета-спирали. Модельные эксперименты показали, что составленная таблица композиционного кода работает без ошибок. Позднее, когда сборка моделей белка была автоматизирована, обнаружился цикл лизоцима, замкнувшийся через дисульфидный мостик без фолдинга, т.е. строго по таблице композиционного кода. Ещё позднее я нашел ещё 5 фрагментов разных лизоцимов, замнувшихся без фолдинга. Более того, в процессе этого исследования я обнаружил замыкание дисульфидных мостиков не только между цистеинами, но и между цистеином и метионином, что явилось научным открытием, согласитесь. Вам известно, что дисульфидные мостики могут образовываться между цистеином и метионином?
Подробнее: http://nanoworld88.narod.ru/data/586.htm Kushelev: Aest, меняйте согласованно углы. Но не забывайте, что эти углы не лежат в одной плоскости aest: Возьмите металлическую цепь о тысячи звеньях, замкните. Бросьте комком на землю. Сколько там углов между звеньями согласованно изменились не лежа при этом в одной плоскости? Цепь не разомкнулась. Мистика... Кушелев: Жаль, что Вы не отличаете механизм белковой молекулы от механизма цепи. В молекуле белка вращение происходит вокруг некоторых осей:
В учебнике молекулярной биологии есть такой термин: "вращение затоможено". Это определяется как раз структурой электронной оболочки молекулы. Так же, как рука может гнуться в локте не по любому направлению. Сустав - это не шарнир Кушелев: Безусловно есть цепи, где звенья не болтаются, как шарнир. Но это не означает, что они согласованы именно так, как в молекуле белка. То, что для Вас очевидно, может быть банальной ошибкой по теме школьной геометрии Я Вам и на примере спиралей белка показал, что фолдинг вторичной структуры - мистика. Спирали формируются рибосомой сразу по таблице композиционного кода. Их структура фиксируется водородными связями в 100 раз быстрее, чем добавляются новые аминокислотные остатки. Мне даже удалось смоделировать процесс трансляции: http://nanoworld88.narod.ru/data/240.htm
Теперь мы знаем, как реализована таблица композиционного кода в механизме трансляции белка. aest: изменение углов согласовано, чтобы не разрушить мостик Кушелев: Сказать - одно, а сделать - другое. Или Вы словами строите?
"Я припарковалась правильно" Виктория Соколик: Приведите пожалуйста значения коэффициентов корреляции и их достоверности в следующем сообщении с указанием объема анализируемой выборки. Кушелев: Уважаемая Виктория! Вы же меня по этой научной статье нашли
Программа Пикотех показывает, что первый белок состоит преимущественно из пи-спиралей, а второй - преимущественно из альфа-310-спиралей. Рентгеноструктурный анализ высокого разрешения не может отличить пи-спираль от альфа-310-спирали. На схемах Черезова оба белка альфа-спиральные. Теперь по поводу образования водородных связей в канале рибосомы... Вы согласны, что в канале рибосомы присутствуют протоны? Если согласны, то что им мешает через миллисекунду после установки очередного аминокислотного остатка образовать водородную связь? Т.е. ещё до того, как тРНК отпустит аминокислотный остаток, который она держит 4-мя вандерваальсовыми связями:
 Подробнее: http://nanoworld88.narod.ru/data/216.htm
Подробнее: http://nanoworld88.narod.ru/data/247.htm aest: Кто ошибку замечает, но не предлагает заменить треугольник на многоугольник с бОльшим количеством углов (как в реальном белке), тому научная истина не интересна А Вы предложили. Только не подумали о том, что параллелограмм не соответствует 3D структуре белка. Сейчас включим ещё один уровень нейронных сетей, и придём к тому, что изменение углов в 3D модели либо разомкнет дисульфидный мостик, либо сломает механизм белковой молекулы Вы поняли, про какой угол мы с Викторией дискутируем? Он отсчитывается вокруг этой вертикальной оси:
И для каждой аминокислоты (в разных спиралях белка) эта ось (ось вращения tRNA) наклонена по-своему... Для 310-спирали и альфа-спирали угол поворота tRNA отличается на 120-100=20 градусов. Среднее значение примем за начало отсчета, т.е. 0 градусов. Пи-спираль строится, если аминокислота повернута на 120 градусов от начала отсчета (от альфа-310), а бета-спираль строится, если аминокислота повернута на 240. Время установки аминокислот отличается. Для альфа-спирали оно минимально, для пи-спирали раза в два больше, а для бета-спирали еще раза в два больше. Поэтому сборка бета-спирали идет примерно в 4 раза медленнее, чем сборка альфа-спирали. Кстати, угол поворота тРНК не совпадает с углом между остатками аминокислот в альфа-, 310-, пи-, бета-спиралях. Поэтому углы поворота tRNA отличаются на 120 градусов, а углы в белковых спиралях, т.е. уже вокруг оси симметрии белковых спиралей получаются ~120 для 310-спирали, ~100 для альфа-спирали, ~87 для пи-спирали и больше 180 для бета спирали. Это - чистая геометрия, но ... не на плоскости
Почему у Виктории получились белковые спирали с углами 0, 120 и 240 градусов при отсчёте вокруг оси белковой спирали? А по той простой причине, что она использовала плоскую схему вместо пространственной модели:
А на плоской схеме тетраэдрический угол в 109 градусов и 28 минут не получится... И что делать? Нарисовать угол 120 градусов, а потом "дофолдить" его до правильного угла альфа-спирали (100 градусов), пи-спирали (87 градусов) или бета-спирали, которая по плоскому чертежу получается вообще не спираль, т.к. там угол 0 градусов Вы пробовали строить спираль с углом вокруг оси симметрии спирали в 0 градусов? Попробуйте. Виктория утверждает, что у неё получилось!
 Но тут каждая следующая аминокислота повёрнута на 180 (не путать с нулём!) градусов. И это, кстати, ближе к реальности. Реальный угол между аминокислотными остатками в бета спирали больше 180 градусов, но меньше 270. А если строить бета-спираль или "тяж", как его любят называть профессионалы, по плоской схеме от Виктории Соколик, то аминокислотные остатки не нужно поворачивать на 180 градусов. Ведь в таблице Виктории однозначно написано, 0 градусов, а никакие не 180. Это значит, что аминокислотные остатки в бета-"тяже" нужно размещать параллельно друг другу, т.е. путём параллельного переноса на шаг одного остатка. "И это правильно", - утверждает Виктория. Или неправильно? *** Kushelev: в белке оси, вокруг которых вращаются аминокислотные остатки не параллельны...aest: Так параллелограмм просто показывает принцип. Кушелев: Ну-ну. Принцип тетраэдричности на примере параллельности. Такие штуки до добра не доводят. У Виктории, например, по плоской схеме получились спирали с углами 0, 120 и -120 градусов. А таких спиралей в природе только одна 310-спираль. И то приближенно. На самом деле и 310-спираль имеет другой угол. Цитата: 310- и пи-спирали содержащие соответственно 3 и 4,4 аминокислотных остатка на 1 виток Кушелев: В инете для пи-спирали встречается два числа. 4.1 остатка на виток и 4.4. Какое из значений правильное? число 3 для 310-спирали тоже нужно уточнить. Но это можно сделать только на достаточно длинных 310-спиралях. А официально считается, что длинных 310-спиралей не бывает. Это совсем недавно мне удалось обнаружить 310-спираль длиной более 1000 витков. Учитывая, что спирали белков упругие, понятно, почему для альфа-спирали пишут значение 3.6 ... 3.7 аминокислотных остатка на виток. Хотя в последнее время уже упростили до 3.6. Понятно, что и для 310-спирали на самом деле есть некий интервал типа 2.95 ... 3.05 аминокислотных остатка на виток. Это же относится и к пи-спиралям, и к бета-спиралям. Итак, углы 0, а точнее -10 для альфа-спирали и +10 для 310-спирали, 120 для пи-спирали и 240 для бета-спирали соответствуют совсем другим углам, если их отсчитывать вокруг оси симметрии белковых спиралей. Для 310-спирали это получается примерно 120 градусов, для альфа-спирали примерно 100 градусов, где-то я встречал значения 97...100 градусов, для пи-спирали 87 градусов, что соответствует 4.1 ... 4.2 остатка на виток. Для бета-спирали официально считается 180 градусов, что тоже нужно уточнять... На самом деле после настройки углов 310-, альфа- и пи-спиралей угол бета-спирали "лежит на поверхности". В бета-спирали на виток получается где-то 1.5 АО, а угол относительно оси спирали 240 градусов соответственно. Виктория Соколик: за правильными цитатами завуалированы умышленно НЕПРАВИЛЬНЫЕ трактовки Кушелева, которые он активно мне приписывает. Кушелев: Ну так поправьте. Как Вы считате, вращение аминокислот на углы 0, 120, 240 градусов соответствует таким же углам между аминокислотными остатками в альфа-, 310-, пи-, бета-спиралях или другим?
 Судя по этой плоской схеме Вы эти углы не отличаете друг от друга...
Кушелев: Фолдинг у Виктории, как я понял, это перевод углов из одной системы отсчета (ось тРНК) в другую систему отсчета (ось белковой спирали) Но это кажущийся фолдинг, согласитесь. Вы поворачиваете аминокислоту вокруг одной оси, потом наклоняете эту ось и наблюдаете другой угол. Так же как с тетраэдром. Поверните тетраэдр вокруг вертикальной оси на 120 градусов, а потом наклоните, чтобы посмотреть вдоль ребра. И угол в 120 градусов "сфолдирует" в вашем сознании (но не в реальном мире) в угол 109 градусов 28 минут
Увжаемая Виктория! А Вас не смутил угол 0 градусов? Или Вы его "смело заменили" на 180 градусов? Кстати, эту ошибку легко исправить. Достаточно организовать "фолдинг" таблицы композиционных углов от Виктории Соколик, чтобы получилась таблица композиционных углов от Александра Кушелева. Сами структуры белков фолдировать не нужно. Достаточно фолдировать таблицу углов А я никак не мог понять, откуда Вы такие углы берете? Это можно назвать "антифолдинг" таблицы композиционных углов

Получается, что Вы не показывали Prosolver иллюстрации, где показано, вокруг каких осей вращаются модели аминокислотных остатков?
Есть же описание алгоритма от Евгения Неделько, которое находится в открытом доступе с 1998 года. В программе Неделько выводились только координаты центров атомов, а углы были взяты у Рамачандрана. Это уже в более поздней версии Дениса Савина программа скрипт стала выводить не только координаты центров атомов, но и кольцегранные оболочки. Помню Вы там ещё ошибку нашли. Правда, программа правильно заработала после исправления второй ошибки. Оказалось, что Денис Савин перепутал в одном месте координату в уравнениях. Потом мы с Валентином поменяли зеркальные отражения аминокислотных остатков на правильные и промасштабировали модели по экспериментальным данным. Кстати, в программе Дениса Савина вращение тоже реализовано не вокруг одной оси, как было бы естественно, а последовательно вокруг трёх декартовых осей. Это уже связано с вычислительной средой. При этом сначала оказалось, что все спирали строятся правильно, а изломы спиралей неправильно. Выяснилось, что правильные углы между спиралями разных типов получаются при правильном задании начала отсчета углов. Другими словами, нужно поставить аминокислоту в правильную позицию, правильно сориентировать по всем трем осям, после чего получаются корректные модели белков. Но это только геометрический уровень. Нужно ещё учесть транспозиционные углы, которые отличаются в разных спиралях и зафиксированы водородными связями. Met и Pro - отдельная "песня". Виктория Соколик: В монографии с картинками показаны оси вращения и точки отсчета углов, которые реализованы в моей программе. Я умышлено не шла по Вашему пути трех осей и углов для них, чтобы не повторять Ваших заблуждений. Читайте матчасть. Кстати Prosolver здесь совсем не причем, а программу Молекулярный конструктор написал Сергей Мельник по моему алгоритму. Кушелев: поворот аминокислот в рибосоме и при вращении белковых спиралей происходит вокруг разных осей. Поэтому набору углов 0, 120, 240, которые отсчитываются вокруг оси вращения тРНК соответствуют углы 97..100, 119...121, ~87 и ~240 в альфа-, 310-, пи- и бета-спиралях. Чтобы это понять, нужно посмотреть на тетраэдр сверху. Кажется, что между его гранями углы в 120 градусов. Но на самом деле в пространстве эти углы по 109 градусов 28 минут. Почему? А потому что они отмеряются не вокруг вертикальной оси, вдоль которой мы смотрим на тетраэдр сверху, а вокруг оси двухгранного угла, совпадающей с наклонным ребром тетраэдра. Давайте посмотрим, куда Вы "умышленно не шли и куда пришли"...
 Кушелев: В книге "Пикотехнология белков" Вы пишите бета-спираль, а не тяж. Это правильно? Я просто уточняю, т.к. Вы обычно меня поправляете по вопросу "бета-спираль". Из книги "Пикотехнология белков": Случайное чередование R-, 0- и L-ротамеров пептидной связи в полипептидной цепи образует неструктурированный фрагмент белка или выпетливание между мотивами с вторичной структурой (рис. 3.17). Однако протяженные участки левой спирали не только экспериментально не выявлены, но и не коди руются в генах белков. Как правило, можно наблюдать 3-4-х аминокислотные единичные витки левой спирали, которые мы склоны отнести к структуре поворотов, экспериментально уверенно регистрируемых методами ЯМР и РС. Кушелев: Тут интересно проверить утверждение "протяженные участки левой спирали не только экспериментально не выявлены, но и не кодируются в генах белков" Дело в том, что по нашим таблицам композиционного кода эти спирали (левая и правая) имеют длину более 263 витков: Как быть с длинными левыми спиралями, которые показывает Пикотех и Молекулярный конструктор?Перечитываем книгу "Пикотехнология белков"...
Кушелев: Что мы видим на этой схеме? Поворот аминокислот вокруг оси tRNA на 120 градусов. Но оси симметрии белковых спиралей НЕ СОВПАДАЮТ с осью, вокруг которой поворачиваются аминокислоты! Этот факт не лежит на поверхности, но ... именно он превращает значения одних углов (0, 120, 240) в значения ДРУГИХ углов (альфа=97...100, пи=-86...88, бета). Если учесть это преобразование углов, то вместо неправильных 0, 120, 240 получатся правильные углы альфа-, бета-, пи-спиралей, которые не деформированы и не зафиксированы водородными связями. Если же учесть водородные связи, то альфа-угол будет изменен в альфа-спирали в одну сторону, а в 310-спирали в другую примерно на 10 градусов. Изменен он будет и в пи-спирали. Тоже на несколько градусов. Изменение коснётся не только композиционных, но и транспозиционных углов. Если это учесть, то геометрический алгоритм поможет получить более точную 3D модель. Учесть нужно и изменение в первую очередь транспозиционного угла в случае Met. Учет изменение третьего угла (угла Pro) даст дополнительную гибкость модели на остатках Pro. Это всё - чистая геометрия. Далее начнётся учет физико-химических взаимодействий. Но делать это нужно с учетом кольцевой формы электрона и соответственно кольцегранной формы аминокислотных остатков. А теперь попробуем найти то место в рассуждениях, где вкралась эта фатальная ошибка. Виктория Соколик: В монографии с картинками показаны оси вращения и точки отсчета углов, которые реализованы в моей программе. Я умышлено не шла по Вашему пути трех осей и углов для них, чтобы не повторять Ваших заблуждений. Читайте матчасть. Кушелев:
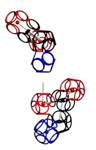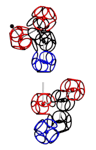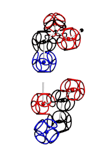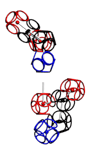 Аминокислота поворачивается вокруг вертикальной оси (оси симметрии tRNA)
 Поворот на 120 градусов относительно оси симметрии tRNA формирует углы альфа-310-, бета-, пи-спиралей соответственно. Подробнее: http://nanoworld88.narod.ru/data/220.htm Технология трансляции генетического кода в структуру белка: http://nanoworld88.narod.ru/data/227.htm Виктория докладывала о кодировании углов поворота аминокислот:
 Подробнее: http://nanoworld88.narod.ru/data/231.htm
Казалось бы, что Виктория пишет всё правильно:
На уровне логики всё верно, если не делать тонких отличий 310-спирали от альфа-спирали.
А здесь и на геометрическом уровне всё правильно! Карты Рамачандрана... Это карты углов поворота следующего аминокислотного остатка относительно предыдущего. Вокруг этой самой оси:
Казалось бы, что Виктория всё понимает, правильно докладывает, показывает правильные картинки, называет правильные карты Рамачандрана...
 И тут, вдруг, бац! И после правильных картинок в верхнем ряду идёт ... нижний ряд, где показана другая ось, т.е. не ось вращения tRNA, не ось вращение аминокислоты относительно соседней, а ось белковой спирали. А углы ... антифолдировались! Как же я не заметил раньше? Просто не мог даже представить ошибку такого уровня...
Это всё равно, что рулить в другой плоскости Смотрим ещё раз внимательно:
 Здесь Виктория показывает вращение вокруг оси симметрии tRNA, т.е. правильно.
 А здесь Виктория поменяла ось симметрии tRNA на ось симметрии белковой спирали. При этом смена системы координатных осей автоматически приводит к изменению углов, т.е. это преобразования координат при переходе в другую систему отсчета, а вовсе не фолдинг!
Создан онлайн сервис "Определение структуры белка по нуклеотидной последовательности" Kushelev's PicoShop Проект для Бумстартера
На базе научного открытия нами создан онлайн-сервис по определению структуры белковых молекул. Теперь мы сможем зарабатывать вместе. По старой технологии определение одной структуры белка обходится примерно в 10 000 евро, а ждать нужно от 2 месяцев до 3 лет. По новой технологии структура определяется в 1000 раз точнее и в миллиард раз быстрее. 80% от найденного Вами заказа принадлежат Вам, как менеджеру. Наш лозунг: "В 1000 раз лучше, в 1000^3 быстрее и в 1000 раз дешевле!" Ваша задача заключается в размещении рекламы на онлайн-сервис белковых структур. Рынок этих структур очень большой и продолжает стремительно расти. Ежедневно кто-то оплачивает до 60 структур по средней цене 10 000 евро за штуку. Новая технология позволяет на одном персональном компьютере обслужить до 100 заказчиков в день. При этом качество результата, полученного по новой технологии в 1000 раз выше по точности, в миллиард раз по быстродействию и в 30 раз шире по номенклатуре белковых молекул. Единственное, что нам сегодня не хватает - рекламы. Как получить Вашу первую зарплату менеджера? Найти заказчика белковых структур и убедить его заказать за счёт лаборатории Наномир пробный заказ. Когда заказчик распробует новую технологию, он начнёт делать коммерческие заказы. С первого коммерческого заказа менеджер получает 80%. С последующих заказов процент будет постепенно уменьшаться, но с первого заказа другого заказчика менеджер снова получит 80%. Зарплата менеджера может достичь миллиона евро в день. И это не предел.
Первая 1000 белковых структур за счёт лаборатории Наномир (на 10 миллионов евро по старой технологии). Инвестирование научных проектов Приглашаем инвесторов и меценатов. Все это реально создать в ближайший год-два при наличии достаточного финансирования. 1. Online service PROTEIN PICOTECHNOLOGY 2. Сверхдобротные одномодовые диэлектрические резонаторы в т.ч. с большим диапазоном перестройки 3. Станки для производства высокодобротных одномодовых резонаторов 4. Технология изготовления сапфировых линз 5. Магнитный тороидально-сферический конструктор 01 Ruby Emdrive (Микроволновый двигатель без реактивной струи) 02 Ruby Power Source (Микроволновый источник энергии) 03 Средство продления жизни (Возвращение молодости) 05 Шестеренчатая передача Кушелева 06 Магнитный подвес-стыковка-герметизация модулей 07 Ионно-микроволновый фрактальный излучатель 08 Гибкий отражатель из жестких элементов 11 Конструктивные компьютерные игры 12 Интеллектуальный кодовый замок 15 Программируемая архитектура 16 Источник энергии промышленной частоты 17 Источник энергии постоянного тока 18 Монокристаллическая видеокамера 19 Система определения активных участков белка 20 Тераваттный лазер непрерывного действия 21 Бактериальный синтез алмазов 22 Шестеренчатые передачи с тремя степенями свободы 24 Безосевая шестеренчатая передача 25 Aктивный язык программирования 26 Телевидение миллиметрового и оптического диапазонов 28 Компьютерный экран из автономных элементов 30 Сверхсветовая локация / зрение 31 Нейтрализатор акустического сигнала Коммерческое предложение: Виктория Соколик: Уважаемые коллеги, Вашему вниманию предоставляется услуга -- моделирование 2D и 3D структуры любого белка без ограничений в его размере и степени изученности с помощью программного обеспечения, базирующемся на принципиально новом подходе декодирования нуклеотидной последовательности, детерминирующей данный белок. Всё, что необходимо от заказчика, это нуклеотидная последовательность мРНК интересующего его белка (или код этой нуклеотидной последовательности в EMBL, или хотя бы код самого белка в PDB). В течение 1-3 суток мы готовы предоставить Вам схему вторичной структуры заказанного белка (2D), модель его пространственной структуры (3D) в виртуальном пространстве, а также файл .pdb с координатами каждого атома белка. Файл .pdb может быть использован по аналогии с файлами закристаллизованных белков из PDB банка для дальнейшего конформационного анализа белка методами молекулярной динамики с учётом физико-химической специфики микроокружения белка или его взаимодействия с лигандами. Таким образом, Вы сможете максимально быстро удобным для Вас способом (по электронной почте, на сайте либо на электронном носителе) получить информацию о структурном шаблоне Вашего белка. Сотрудничество может быть различным: - участие в научных дискуссиях на форуме (конструктивное) - совместное создание коммерческого продукта - поиск инвесторов - выступить менеджером по продаже готовых коммерческих продуктов - конструктивные предложения по продвижению идей лаборатории Наномир - содействие в проведении экспериментов и т.п. - написание совместных научных статей и т.п. - материальный
вклад (денежный или обеспечение оборудованием и материалами)
Пожалуйста, сообщайте о своем вкладе, чтобы мы зачли Вас как партнера лаборатории Наномир. +7-926-5101703, +7-903-2003424, +7-916-8265031, Skype: Kushelev2009, mail: kushelev20120@yandex.ru веб-мани: WM-кошелек R426964799301 Другие способы перевода Огромное спасибо всем за помощь и поддержку! |
| В избранное | ||